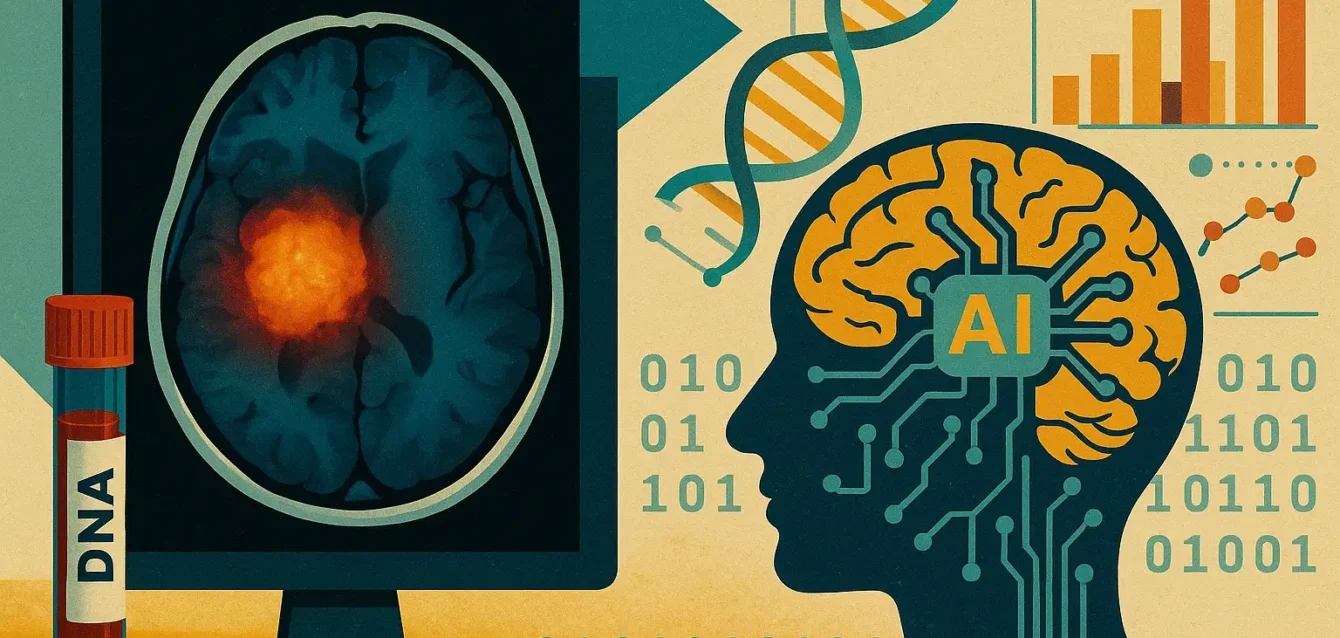
Una gota de sangre que reemplaza al bisturí: la IA que cambia el diagnóstico de tumores cerebrales
Del laboratorio al paciente: cómo funciona y por qué importa
Hasta hoy, descubrir un tumor en el cerebro implicaba un itinerario casi invariable: resonancia magnética para localizar la lesión, ingreso al quirófano, apertura del cráneo, extracción de una porción diminuta de tejido y, finalmente, varios días de espera mientras un patólogo confirmaba el resultado al microscopio. Todo ese recorrido era el peaje obligatorio para saber con qué clase de cáncer se enfrentaban médicos y paciente. La nueva herramienta conocida como Cross NN propone un atajo que parece sacado de la ciencia-ficción, pero ya está validado: toma una muestra de sangre del brazo, detecta en ella fragmentos de ADN liberados por el tumor y, con ayuda de inteligencia artificial, entrega un diagnóstico con más del 99 % de exactitud sin que nadie levante un bisturí.
Para entender el salto conviene imaginar al ADN tumoral circulante como confeti microscópico que el cáncer va soltando a la corriente sanguínea. Esos trozos de material genético no viajan desnudos: llevan pequeñas “etiquetas” químicas llamadas metilaciones, que funcionan como un código de barras capaz de señalar, con sorprendente detalle, la identidad del tumor. El análisis empieza en un laboratorio de biología molecular donde se separa ese ADN libre; luego interviene un software entrenado con miles de huellas moleculares de tumores ya conocidos. La red neuronal compara el patrón de etiquetas del paciente con su biblioteca y devuelve una respuesta: qué tipo de tumor es, cuán agresivo suele ser y, a menudo, qué tratamientos han demostrado mejor resultado en casos similares. El informe llega al oncólogo en cuestión de horas.
En términos prácticos, la diferencia para el paciente es enorme. En lugar de prepararse para una intervención que exige hospitalización, anestesia y un periodo de recuperación lleno de posibles complicaciones, solo necesita acudir a un servicio de extracción venosa y volver a casa. Tampoco hay que vivir la semana de incertidumbre que suele acompañar a una biopsia tradicional: el resultado está disponible en dos días y permite iniciar la estrategia terapéutica sin demoras. Es, literalmente, cambiar una cicatriz en la cabeza por una pequeña tirita en el brazo.
Los beneficios se extienden al equipo médico. Con información molecular precisa al principio del camino, el neurocirujano decide si conviene intervenir para extirpar la mayor parte del tumor o si, por su ubicación y tipo, resulta más sensato comenzar con radioterapia, quimioterapia o inmunoterapia. Cuando la cirugía sí aporta ventaja —por ejemplo, para descomprimir áreas críticas o reducir masa tumoral antes de irradiar—, entra el bisturí, pero ahora respaldado por un mapa genético que facilita planificar hasta dónde cortar y qué recuperar. El tiempo que antes se empleaba en esperar resultados histológicos se convierte en tiempo de tratamiento, y eso, en oncología, suele traducirse en mayor supervivencia.
Para los hospitales, la adopción del test significa reordenar recursos. Menos cirugías puramente diagnósticas liberan quirófanos y camas de cuidados intensivos; se reducen los días de estancia posoperatoria y las complicaciones asociadas a la manipulación de tejido cerebral. La inversión se desplaza hacia laboratorios de secuenciación y profesionales capaces de interpretar informes genómicos, pero diversos estudios de costo-efectividad sugieren que el balance global es favorable: cada paciente que evita una biopsia ahorra gastos de quirófano, reduce riesgo de readmisión y, sobre todo, gana calidad de vida.
Ahora bien, ninguna innovación está exenta de retos. Uno clave es la igualdad de acceso. Equipar un laboratorio con secuenciadores de última generación cuesta dinero, y existe el riesgo de que solo los grandes centros urbanos ofrezcan la prueba mientras hospitales rurales o países con menos recursos sigan dependiendo del bisturí. Para evitarlo, varios sistemas sanitarios prueban modelos de laboratorio centralizado: hospitales comarcales extraen la sangre y la envían refrigerada a un centro de referencia que procesa las muestras en lotes, abarata costes y devuelve los datos por vía digital. Al mismo tiempo, fabricantes de tecnología trabajan en equipos de mesa más económicos que podrían democratizar la secuenciación avanzada.
Otro frente es el de la privacidad genética. El ADN tumoral circulante revela detalles sobre el cáncer, pero también puede exponer mutaciones heredables que el paciente quizás no quiera conocer. Por eso, las instituciones incorporan protocolos de consentimiento informado donde se explica qué se analiza, qué podría descubrirse y qué opciones tiene la persona en caso de hallazgos inesperados. Además, los datos se almacenan en servidores cifrados y se anonimiza la información antes de usarla para mejorar el algoritmo, de modo que nadie pueda rastrear un resultado hasta un individuo concreto sin la debida autorización.
Con todo, los especialistas señalan que el mayor valor de esta tecnología no está solo en lograr un diagnóstico sin bisturí, sino en lo que ocurre después. Al repetir el test cada pocas semanas, los médicos pueden vigilar cómo cambia la “firma” molecular del tumor con el tratamiento. Si los fragmentos de ADN disminuyen, la terapia funciona; si aumentan o surgen nuevas etiquetas químicas, es señal de que el cáncer intenta escapar y hay que cambiar de estrategia antes de que aparezcan síntomas o la resonancia muestre progresión. En otras palabras, la misma gota de sangre que reemplaza la biopsia sirve para ajustar la medicación en tiempo real y personalizar el cuidado hasta un nivel jamás visto.
La comunidad científica ya investiga cómo llevar este método a otros cánceres: pulmón, páncreas, mama, colon. La promesa es que, en un futuro no tan lejano, un chequeo anual de sangre pueda detectar varios tumores en fases tan tempranas que la intervención sea mínima y las oportunidades de curación se disparen. Mientras tanto, la neuro-oncología vive su propio renacimiento: la cirugía se reserva para tratar, la sangre para diagnosticar y el algoritmo para guiar el camino.
El gran desafío ahora es integrar esta revolución sin dejar a nadie atrás, formar médicos que hablen tanto el lenguaje de la biología molecular como el de la empatía, y establecer reglas claras que protejan la privacidad genética. Si se logra ese equilibrio, la historia clínica de los tumores cerebrales pronto se escribirá sin cicatrices: bastará una jeringa, un laboratorio y un modelo de inteligencia artificial dispuesto a escuchar lo que el cáncer confiesa en cada gota de sangre.
Seguirle el rastro al tumor en tiempo real
Del protocolo hospitalario a la práctica diaria
La primera gran promesa de la biopsia líquida epigenética –saber qué tumor hay en el cerebro sin abrir el cráneo– ya se está cumpliendo en centros de referencia. El siguiente objetivo es aún más ambicioso: usar la misma gota de sangre para vigilar, casi día a día, cómo reacciona el cáncer a cada fármaco y ajustar la terapia antes de que la enfermedad gane ventaja. En la práctica, esto exige un engranaje clínico que combine rapidez de laboratorio, potencia de cálculo y una nueva cultura de decisión médica basada en datos que llegan sin pausa.
El circuito empieza cuando el equipo de neuro-oncología recibe el diagnóstico molecular inicial. Esa “foto” del tumor incluye la identidad epigenética, las mutaciones dominantes y, sobre todo, las rutas de señalización que sostienen su crecimiento. Con esa hoja de ruta, el oncólogo prescribe un esquema de tratamiento lo más ajustado posible: quimioterapia clásica si la metilación de MGMT indica buena respuesta a alquilantes, un inhibidor de tirosina-quinasa si el informe revela amplificación de EGFR, o combinación de inmunoterapia y antiangiogénicos si la firma epigenética sugiere un microambiente tumoral infiltrado por linfocitos. El día en que se inicia el fármaco, el paciente entrega la primera muestra de control: servirá de línea base para todo el seguimiento.
Dos semanas después, antes de que los efectos adversos se vuelvan intensos, llega el segundo pinchazo. El laboratorio repite el protocolo de extracción, bisulfitación y secuenciación; la inteligencia artificial compara la nueva curva de metilación con la inicial y emite un informe de variación. Cuando la carga total de ADN tumoral circulante cae de forma sostenida, la plataforma asigna un color verde: el tratamiento parece funcionar y el plan continúa sin cambios. Si la curva se aplana o –peor aún– sube de forma abrupta, salta una alerta amarilla o roja que sugiere reevaluar. El oncólogo, con esa señal, decide adelantar la resonancia o cambiar de línea terapéutica sin esperar a que el tumor crezca lo suficiente para hacerse notar en la imagen.
En el hospital universitario de Heidelberg, uno de los primeros en implementar el seguimiento seriado, se documentó un caso ilustrativo: una mujer de 42 años con oligodendroglioma de bajo grado empezó temozolomida y radioterapia fraccionada. A las cuatro semanas, el test epigenético mostró una caída del 65 % en la carga de ADN tumoral; la resonancia seguía casi idéntica, pero el equipo mantuvo el esquema confiado en la señal molecular. Tres meses más tarde, la imagen confirmó reducción volumétrica. En contraste, otro paciente con glioblastoma progresivo exhibió un alza súbita de metilaciones en regiones reguladas por genes de reparación de ADN después del primer ciclo de quimio. El sistema recomendó un inhibidor de PARP; la carga bajó y la progresión clínica se aplazó cinco meses, un lapso valioso en un tumor notoriamente agresivo.
Para que estos virajes terapéuticos sean factibles, el laboratorio debe entregar resultados en un máximo de 48 horas. Esa velocidad obliga a automatizar pasos que antes requerían intervención manual. Secuenciadores de mesa con flujos de casete sellado permiten cargar la muestra a las ocho de la mañana y tener listas las lecturas brutas al final de la tarde. Un servidor local pre-procesa las secuencias y, tras una capa de cifrado, las envía al clúster central que ejecuta el algoritmo. El informe regresa firmado digitalmente al oncólogo jefe antes de la siguiente ronda matutina de visitas. Si el centro carece de infraestructura, la muestra viaja por mensajería a un hub nacional que replica el mismo flujo nocturno; la región de Lombardía, en Italia, ya coordinó nueve hospitales comarcales que operan bajo ese modelo y mantienen el plazo de dos días.
La infraestructura es solo la mitad de la ecuación. La otra mitad es cultural. El personal clínico está habituado a decisiones ancladas en imágenes radiológicas y biopsias fijas; ahora debe asimilar gráficos que se mueven como cotizaciones bursátiles y aprender a interpretar variaciones pequeñas pero relevantes. Programas de educación continua enseñan a leer curvas de carga tumoral, a correlacionar picos y valles con perfiles de resistencia y a comunicar al paciente que un cambio molecular puede ser más importante que la foto estática de la resonancia.
En términos de coste-efectividad, la monitorización seriada aún se halla en periodo de evaluación, pero los primeros números son alentadores. Cada ciclo de quimioterapia que se suspende a tiempo ahorra miles de euros en fármacos y neutraliza costes de hospitalización por toxicidad. Las aseguradoras privadas de los Países Bajos acordaron reembolsar tres controles epigenéticos por paciente y por línea de tratamiento bajo la premisa de que reducirán al menos un 20 % el gasto total en oncología neuro-quirúrgica. En sistemas públicos, como el británico, se explora un modelo de copago cero cuando el algoritmo evita una segunda cirugía no curativa.
Queda un reto científico: definir umbrales universales de respuesta. ¿Basta que la carga de ADN tumoral baje un 20 %? ¿O se necesita un 50 % sostenido? Consorcios de Europa y Norteamérica analizan miles de curvas de seguimiento para establecer criterios que funcionen en gliomas, meduloblastomas y metástasis. Mientras, se emplea la regla práctica “dos mediciones consecutivas a la baja” como indicador provisional de buena evolución.
No conviene olvidar la dimensión humana. El paciente vive estos análisis seriados con emociones encontradas: alivio porque evita cirugías y ansiedad porque un simple cambio de color en la hoja de resultados puede indicar recaída. Aquí, la consulta con el oncólogo o la enfermera de referencia cobra un valor enorme: explicar con calma qué significa cada curva, qué márgenes de error existen y qué pasos siguen si aparece una alerta. Esa contención hace que la tecnología no se sienta fría, sino empática.
Por último, la expansión a otros tumores cerebrales poco frecuentes, como los del tronco encefálico o los embrionarios en niños, exige que la biblioteca epigenética incluya firmas raras. Gracias al aprendizaje federado, hospitales de Brasil, Sudáfrica y la India ya envían datos anonimizados para alimentar la red, lo que incrementa la precisión global y garantiza que un niño de Quito o una adulta de Yakarta reciban el mismo nivel de exactitud que un paciente de Boston o Múnich.
La monitorización en tiempo real es, así, la segunda etapa natural tras el diagnóstico sin bisturí. La sangre no solo nombra al tumor; lo vigila, lo delata cuando muta y ayuda a cambiar de estrategia antes de que la enfermedad muestre su peor cara. Con cada tubo extraído, Cross NN y su descendencia algorítmica consolidan un camino en el que la medicina se vuelve dialogante: el paciente aporta muestras mínimas, la máquina traduce sus mensajes y el médico convierte datos en decisiones. Si el cáncer cerebral era un terreno en el que cada avance se pagaba con cirugía y meses de espera, hoy parece inclinarse ante una nueva lógica donde la información molecular corre más rápido que la enfermedad. Ese giro, ya irreversible, redefine la manera de entender la esperanza: no como una fe ciega en lo que el bisturí pueda encontrar, sino como la confianza fundada en la capacidad de leer el tumor a cada paso y anticiparse a sus próximos movimientos.
De la innovación al estándar: qué falta para que la biopsia líquida epigenética sea la norma
La prueba basada en Cross NN ya demostró que puede reemplazar a la biopsia quirúrgica y, además, vigilar al tumor en tiempo real. Sin embargo, transformar un hallazgo de laboratorio en práctica universal exige superar varios obstáculos concretos. El primero es regulatorio. Las agencias de medicamentos deben evaluar no solo la precisión de la prueba sino también su reproducibilidad entre laboratorios; por eso, la Red Europea de Neuro-oncología y la FDA estadounidense exigen estudios multicéntricos que incluyan hospitales de distinto tamaño, equipos de secuenciación heterogéneos y personal con grados variados de experiencia. Los datos iniciales son sólidos: la concordancia interlaboratorio supera el 97 %, pero faltan ensayos de implementación “en el mundo real”, donde las muestras no siempre llegan en condiciones ideales y los pacientes presentan comorbilidades que pueden complicar la interpretación.
El segundo obstáculo es financiero. Un test de secuenciación profunda cuesta, hoy, entre 400 y 600 euros si se procesa en lotes grandes; la cifra puede subir a mil si el hospital maneja pocas muestras. Aun así, los análisis de costo-beneficio señalan que cada biopsia quirúrgica evitada ahorra, de media, 6500 euros en quirófano, anestesia, cuidados intensivos y gestión de complicaciones. El Servicio Nacional de Salud británico propone un modelo de transición: cubrir al cien por cien la prueba en tumores cerebrales profundos, donde la biopsia es muy arriesgada, e ir ampliando el reembolso a lesiones más accesibles cuando el volumen de muestras abarate todavía más el test.
Un tercer reto, menos visible pero crucial, es cultural. La práctica oncológica se formó durante décadas en torno a la histología clásica. Cambiar ese paradigma implica reeducar a especialistas que confían en la imagen al microscopio, no en una tabla de probabilidades epigenéticas. Sociedades médicas de Canadá, Corea y España han creado diplomados breves, entre uno y tres meses, que enseñan a integrar curvas de ADN tumoral circulante en decisiones diarias: cuándo re-irradiar, cuándo suspender quimioterapia, cuándo derivar a un ensayo de terapia génica. Al mismo tiempo, las facultades de medicina incluyen ya rotaciones de bioinformática clínica, para que la próxima generación entre al hospital sabiendo leer datos masivos con naturalidad.
El horizonte: cribado poblacional y medicina preventiva
Una vez que la prueba se estabilice en neuro-oncología, los investigadores planean dar un paso más audaz: utilizar la misma tecnología para detectar tumores cerebrales antes de que aparezcan síntomas. La idea no es descabellada. Estudios piloto en Japón tomaron muestras de sangre de 1200 personas sanas y, en siete casos, encontraron firmas epigenéticas compatibles con glioma de bajo grado. De esos siete, cuatro desarrollaron lesiones visibles en los siguientes dieciocho meses; los otros tres aún se vigilan. Si las cohortes se amplían, podría surgir un programa de cribado selectivo para personas con mutaciones germinales de alto riesgo o con antecedentes familiares. Detectar un glioma en fase preclínica permitiría resecciones quirúrgicas mucho más pequeñas y, en algunos casos, vigilancia activa sin tratamiento agresivo.
El cribado plantea, sin embargo, dilemas éticos delicados. ¿Cómo manejar un resultado “positivo” cuando la lesión aún no se ve en la resonancia? ¿Cuándo intervenir? Para responder, la Unión Europea financia el proyecto PRECISE, que seguirá durante diez años a 25 000 voluntarios con muestreo de sangre anual y resonancia cada tres años. El objetivo es definir puntos de corte seguros: no alarmar de más ni llegar tarde. Toda la información alimentará a Cross NN 2.0, versión entrenada no solo para diagnosticar, sino para calcular velocidad de crecimiento y ventana óptima de intervención.
Mientras tanto, países con menos recursos exploran innovaciones de bajo coste. En la India, un consorcio público-privado desarrolló un kit que concentra la captura de ADN tumoral circulante en una columna de resina de sílice y usa secuenciadores de flujo continuo más económicos que los de lectura masiva. El rendimiento cae ligeramente (la precisión ronda el 96 %), pero el precio baja a 120 dólares por muestra. Esa democratización evita que la brecha tecnológica se traduzca en la cruda estadística de supervivencia: los ensayos iniciales muestran que, incluso con esa precisión, los pacientes ganan entre dos y cinco meses de calidad de vida al evitar cirugías innecesarias y acelerar tratamientos.
En definitiva, Cross NN marca el inicio de una era en la que los tumores cerebrales se diagnosticarán, tratarán y vigilarán sin necesidad de abrir el cráneo. El desafío pasa ahora de la mesa de laboratorio a la de gestión sanitaria: asegurar acceso, formar profesionales y regular la privacidad genética. Si la sociedad está dispuesta a invertir en esa dirección, la promesa se convertirá en norma y la frase “necesitamos una biopsia” pasará a ser la excepción, no la regla, en el mapa del cáncer cerebral.