Existe un universo invisible que bulle de actividad incesante en cada rincón de nuestra existencia, un cosmos en miniatura donde la vida misma se coreografía. Es el mundo de las proteínas, las macromoléculas que lo hacen prácticamente todo en nuestros cuerpos. Actúan como mensajeras, como catalizadoras de reacciones químicas, como defensoras de nuestro sistema inmunitario y como los ladrillos estructurales de nuestras células. Cada una de ellas es una obra de ingeniería nanométrica, una cadena de aminoácidos plegada con una precisión exquisita en una forma tridimensional única. Esa forma, su arquitectura, lo es todo. Define su función. Un ligero error en su plegamiento puede desencadenar enfermedades devastadoras. Y en esta danza de formas moleculares reside el corazón de la medicina moderna.
Durante más de un siglo, el descubrimiento de fármacos ha sido análogo a un juego de cerrajería a una escala inimaginable. Los científicos se han esforzado por encontrar o fabricar llaves moleculares, los fármacos, capaces de encajar en cerraduras biológicas muy específicas, generalmente proteínas, para activarlas, desactivarlas o modular su comportamiento. Una llave que encaja en la cerradura correcta puede detener la replicación de un virus, corregir un desequilibrio metabólico o impedir que una célula cancerosa se divida. El problema es que el número de cerraduras posibles es astronómico y el taller del cerrajero ha operado históricamente con herramientas lentas y costosas, basadas en una combinación de serendipia, cribado masivo y una laboriosa química computacional. Este proceso, un viaje de una década y miles de millones de euros desde la idea inicial hasta la farmacia, ha sido uno de los mayores cuellos de botella del progreso humano.
Ahora, estamos en el umbral de una revolución que promete transformar radicalmente este paradigma. Una nueva forma de inteligencia, la inteligencia artificial generativa, está emergiendo no como una simple herramienta de análisis, sino como una fuente inagotable de imaginación molecular. Hemos visto a estas inteligencias artificiales componer sinfonías, escribir poemas y pintar cuadros con el estilo de los grandes maestros. Su nuevo lienzo, sin embargo, no es el píxel ni la palabra, sino el átomo. Están aprendiendo el lenguaje fundamental de la biología, la gramática del plegamiento de las proteínas, para diseñar desde cero moléculas completamente nuevas, estructuras que la naturaleza jamás ha concebido pero que podrían resolver problemas médicos que nos han acosado durante milenios.
En el epicentro de esta transformación se encuentra una nueva generación de plataformas y modelos computacionales. Una de las más ambiciosas es AIDO, un sistema concebido por la empresa GenBio.ai que aspira a crear un «Organismo Digital Impulsado por IA», una simulación multiescala de la biología humana. Dentro de esta vasta empresa, un módulo especializado llamado StructureDiffusion actúa como un escultor virtuoso. Su material de trabajo no es el mármol ni la arcilla, sino nubes aleatorias de átomos. Su técnica se basa en un concepto elegante y poderoso conocido como modelos de difusión. Estos modelos aprenden a crear orden a partir del caos, a revertir la entropía a nivel molecular para generar, átomo por átomo, las proteínas funcionales del futuro. Este artículo explora esta frontera fascinante, un viaje para comprender cómo la inteligencia artificial está dejando de ser una mera observadora del mundo biológico para convertirse en su arquitecta. Analizaremos la promesa de esta tecnología, el ingenio que la sustenta y las profundas implicaciones que tiene para la ciencia, la medicina y nuestra propia concepción de la vida.

El antiquísimo arte de encontrar una llave para cada cerradura
Para apreciar la magnitud del cambio que la inteligencia artificial está introduciendo, primero debemos comprender la inmensa complejidad del desafío que pretende resolver. La idea de que la forma de una molécula determina su función biológica, conocida como el principio de complementariedad molecular, es la piedra angular de la farmacología. A menudo se explica con la analogía de la «cerradura y la llave». La proteína diana, implicada en una enfermedad, es la cerradura. El fármaco es la llave que debe tener la forma y las propiedades químicas exactas para encajar en esa cerradura y solo en esa, sin abrir accidentalmente otras puertas, lo que provocaría efectos secundarios no deseados.
Pensemos en un juguete infantil con agujeros de diferentes formas, un cubo, una estrella, un círculo, y las piezas correspondientes. Un niño aprende rápidamente que la pieza con forma de estrella solo cabe en el agujero con forma de estrella. La biología opera bajo un principio similar, pero con una complejidad infinitamente mayor. La «cerradura» de una proteína no es un simple agujero, sino una cavidad tridimensional dinámica con contornos, bolsillos y superficies con cargas eléctricas específicas. La «llave» del fármaco debe coincidir con esta topografía con una precisión atómica.
Históricamente, hemos encontrado estas llaves de varias maneras. La primera fue la casualidad. El descubrimiento de la penicilina por Alexander Fleming es el ejemplo paradigmático: una contaminación accidental de un cultivo bacteriano por un moho reveló la existencia de una molécula con potentes propiedades antibióticas. Durante décadas, muchos fármacos surgieron de la observación fortuita o del estudio de remedios tradicionales. El segundo método, más sistemático, es el cribado de alto rendimiento. Consiste en probar decenas de miles, o incluso millones, de compuestos químicos ya existentes de vastas bibliotecas moleculares para ver si alguno, por azar, muestra actividad contra una diana biológica concreta. Es un enfoque de fuerza bruta, equivalente a probar un llavero con millones de llaves en una sola cerradura con la esperanza de que una funcione. Es costoso, lento y, aunque ha producido éxitos, su tasa de aciertos es extremadamente baja.
Con la llegada de la informática, surgió un tercer enfoque: el diseño racional de fármacos. Los científicos empezaron a utilizar ordenadores para visualizar la estructura tridimensional de las proteínas y diseñar manualmente moléculas que pudieran encajar en sus sitios activos. Este método es mucho más dirigido, pero sigue dependiendo en gran medida de la intuición y la experiencia de los químicos, y el espacio de posibles moléculas para sintetizar es tan vasto que explorarlo de esta manera es como intentar navegar por el océano en un bote de remos. El número de moléculas pequeñas y orgánicas que podrían existir teóricamente es superior a , una cifra que supera con creces el número de átomos en el sistema solar.
El resultado de estas limitaciones es un proceso de desarrollo de fármacos extraordinariamente arduo. El camino desde la identificación de una diana terapéutica hasta la aprobación de un nuevo medicamento dura, de media, entre diez y quince años, con un coste que a menudo supera los dos mil millones de euros. La gran mayoría de los candidatos fracasan en algún punto del camino. Este es el gran muro contra el que se ha estrellado la medicina durante décadas, y es precisamente el muro que la inteligencia artificial generativa promete derribar.
La llegada de un nuevo tipo de imaginación
El término «inteligencia artificial» evoca a menudo imágenes de sistemas que analizan datos, reconocen patrones o clasifican información. El software que detecta el correo no deseado, el algoritmo que reconoce caras en las fotografías o el sistema que diagnostica enfermedades a partir de imágenes médicas son ejemplos de IA discriminativa. Su función es aprender a distinguir entre diferentes categorías de datos existentes. Sin embargo, la revolución que estamos presenciando proviene de una rama diferente de este campo: la IA generativa.
Como su nombre indica, la IA generativa no se limita a analizar, sino que crea. Aprende los patrones, las reglas subyacentes y la «gramática» de un conjunto de datos para luego producir artefactos completamente nuevos que se ajustan a esa misma gramática. La mayoría de nosotros ya hemos interactuado con estos sistemas. Cuando le pedimos a un modelo de lenguaje como ChatGPT que escriba un soneto, no está buscando un soneto existente en su memoria; está generando uno nuevo, palabra por palabra, basándose en su comprensión de las reglas de la rima, el metro y el significado. Cuando una herramienta como Midjourney o DALL-E crea una imagen de «un astronauta montando a caballo en Marte al estilo de Monet», está sintetizando una obra visual original a partir de su conocimiento conceptual del impresionismo, los astronautas y la astronomía.
La pregunta que impulsó a los pioneros en el campo de la biología computacional fue electrizante en su simplicidad: si una IA puede aprender el lenguaje de Shakespeare o la estética de Van Gogh, ¿podría aprender el lenguaje de la vida misma? ¿Podría asimilar las leyes de la física y la química que gobiernan cómo las cadenas de aminoácidos se pliegan en estructuras funcionales y luego usar ese conocimiento para imaginar proteínas que nunca antes han existido?
Los primeros intentos de aplicar la IA generativa al diseño molecular utilizaron arquitecturas como las Redes Generativas Antagónicas (GAN) o los Autocodificadores Variacionales (VAE). Estos modelos demostraron ser prometedores, pero a menudo luchaban con la inmensa complejidad de generar estructuras tridimensionales precisas y estables. El gran avance llegó de la mano de una clase de modelos que se había popularizado en el campo de la generación de imágenes por su asombrosa capacidad para producir resultados fotorrealistas: los modelos de difusión. Su enfoque era diferente, contraintuitivo y extraordinariamente poderoso.
Esculpir proteínas a partir del caos: la magia de los modelos de difusión
Para entender el funcionamiento de un modelo de difusión, imaginemos a un escultor que, en lugar de empezar con un bloque de mármol, comienza con una nube informe de polvo de mármol suspendida en el aire. Su tarea es guiar cada partícula de polvo a su posición exacta para que, al final, la majestuosa forma de una estatua emerja de la nada. Suena a magia, pero es una analogía muy cercana a lo que hacen estos algoritmos.
El proceso de entrenamiento de un modelo de difusión consta de dos fases. La primera es el proceso hacia adelante o de «difusión». El modelo toma un dato perfectamente estructurado, como la imagen de un rostro humano o, en nuestro caso, la estructura atómica de una proteína conocida extraída de una base de datos científica. Luego, de forma iterativa, añade una pequeña cantidad de «ruido» o aleatoriedad a los datos. Paso a paso, la imagen del rostro se vuelve más granulada y borrosa, o las posiciones de los átomos de la proteína se desordenan ligeramente. Este proceso continúa hasta que el dato original se ha corrompido por completo, convirtiéndose en puro ruido estadístico, una nube de píxeles aleatorios o un conjunto de coordenadas atómicas sin sentido. Durante esta destrucción controlada, el modelo no está pasivo; está aprendiendo meticulosamente la operación matemática exacta que se necesita en cada paso para añadir ese ruido.
La segunda fase es el proceso inverso o de «denoising», y aquí es donde ocurre la creación. El modelo comienza ahora con una nueva muestra de puro ruido, una nube de átomos completamente aleatoria. Su objetivo es revertir el proceso que acaba de aprender. Basándose en su entrenamiento, predice el pequeño patrón de ruido que se añadió en el último paso del proceso de difusión y lo resta. De una nube caótica, emerge una estructura con un atisbo de orden. Repite este proceso cientos o miles de veces. En cada paso, la estructura se refina, los átomos se mueven a posiciones más plausibles, se forman enlaces químicos estables y las cadenas de aminoácidos comienzan a plegarse. Es un proceso de refinamiento gradual, como una fotografía que emerge lentamente del desenfoque hasta alcanzar una nitidez asombrosa. Al final del proceso inverso, lo que queda es una estructura proteica completamente nueva, coherente, estable y diseñada según los principios fundamentales de la biofísica que el modelo ha asimilado.
La superioridad de este método para el diseño molecular tridimensional radica en su naturaleza iterativa. A diferencia de otros modelos que intentan generar la estructura final de una sola vez, los modelos de difusión la construyen progresivamente. Esto les permite tener en cuenta simultáneamente las interacciones a larga distancia entre diferentes partes de la proteína y las restricciones locales entre átomos vecinos en cada paso del refinamiento. El resultado es un control y una calidad de detalle sin precedentes, la capacidad de esculpir con una precisión casi atómica.
AIDO y StructureDiffusion: la orquesta y su solista virtuoso
Armados con esta poderosa tecnología, centros de investigación y empresas de todo el mundo han comenzado a construir plataformas para aprovechar su potencial. La visión de GenBio.ai con su plataforma AIDO es particularmente ambiciosa. No se trata simplemente de diseñar una molécula aislada, sino de crear un modelo computacional integral de la biología humana, un «Organismo Digital». La idea es que AIDO funcione como una orquesta sinfónica, con diferentes modelos de IA especializados actuando como secciones de instrumentos, cada uno encargado de simular un aspecto diferente de la biología, desde el ADN y el ARN hasta las proteínas, las células y los tejidos.
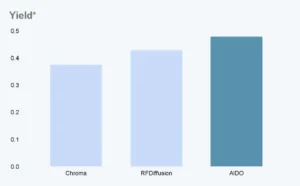
Dentro de esta orquesta, StructureDiffusion es el solista virtuoso, el módulo especializado en el diseño de proteínas mediante modelos de difusión. Su desarrollo se basa en avances fundamentales del mundo académico, como el influyente modelo RFDiffusion de la Universidad de Washington, pero incorpora innovaciones cruciales que lo orientan hacia aplicaciones terapéuticas muy específicas. Su principal ventaja competitiva no es solo la capacidad de generar proteínas válidas, sino de hacerlo con un nivel de control estructural de grano fino que antes era inalcanzable. El científico ya no es un mero espectador que espera a ver qué produce la IA, sino un director que puede imponer condiciones y guiar el proceso creativo con una precisión asombrosa.
Este control se manifiesta de varias maneras. Por ejemplo, los científicos pueden instruir a StructureDiffusion para que diseñe proteínas que se ajusten a una clasificación estructural específica, conocida como jerarquía CAT (Clase, Arquitectura, Topología). Esto es fundamental para construir proteínas complejas con múltiples dominios funcionales, algo esencial para muchas terapias avanzadas. Sin embargo, donde su capacidad de control brilla con más intensidad es en el diseño de una de las clases de proteínas más importantes para la medicina: los anticuerpos.
Los anticuerpos son los centinelas de nuestro sistema inmunitario. Son proteínas en forma de «Y» que pueden reconocer y neutralizarse contra invasores extraños, como virus y bacterias. Su capacidad de unión se concentra en unas pequeñas regiones variables en los extremos de los brazos de la «Y», llamadas Regiones Determinantes de la Complementariedad o CDR. La longitud y la secuencia de estos bucles CDR determinan a qué molécula, o «antígeno», se unirá el anticuerpo. StructureDiffusion permite a los científicos especificar con exactitud características cruciales de los anticuerpos que desean diseñar:
- Pueden definir las longitudes deseadas para los bucles CDR, enfocando el diseño hacia un tipo de diana específico.
- Pueden determinar el tipo de cadena ligera del anticuerpo (kappa o lambda), una característica importante para su producción y estabilidad.
Este nivel de control es revolucionario. Los métodos tradicionales de descubrimiento de anticuerpos a menudo comienzan con la inmunización de animales y luego requieren un arduo proceso de «humanización» para que las moléculas no sean rechazadas por el cuerpo humano. StructureDiffusion evita todo este proceso diseñando desde cero anticuerpos totalmente humanos in silico, es decir, en el ordenador. Es un cambio de paradigma desde la modificación de lo que la naturaleza nos ofrece al diseño intencionado de soluciones biológicas a medida.

La nueva era de los anticuerpos y la medicina personalizada
Las implicaciones prácticas de esta tecnología son inmensas, especialmente en el campo de la inmunoterapia y la medicina personalizada. Los anticuerpos monoclonales, versiones sintéticas de estas proteínas inmunitarias, ya se encuentran entre los fármacos más eficaces y vendidos del mundo, utilizados para tratar desde el cáncer hasta las enfermedades autoinmunes. Sin embargo, su desarrollo sigue siendo un proceso lento y plagado de incertidumbre. La capacidad de diseñar anticuerpos de novo con StructureDiffusion aborda directamente estos cuellos de botella.
Permite una velocidad sin precedentes. En lugar de meses o años de trabajo en el laboratorio para generar y seleccionar anticuerpos candidatos, un modelo de IA puede generar miles de diseños prometedores en cuestión de horas o días. Esto es particularmente crucial en el contexto de una pandemia. La capacidad de obtener la secuencia de un nuevo virus y, en semanas, diseñar in silico anticuerpos neutralizantes de alta afinidad podría transformar nuestra capacidad de respuesta ante futuras amenazas sanitarias globales.
Además, abre la puerta a una medicina verdaderamente personalizada. El cáncer, por ejemplo, no es una única enfermedad. Un tumor en un paciente puede estar impulsado por un conjunto de mutaciones proteicas único. Con el diseño computacional, se vuelve concebible la idea de secuenciar el tumor de un paciente, identificar sus «cerraduras» moleculares únicas y diseñar un anticuerpo a medida para atacarlas específicamente. Esto maximizaría la eficacia del tratamiento y minimizaría los daños a las células sanas, llevando la promesa de la oncología de precisión a un nuevo nivel.
Esta tecnología también permite a los científicos explorar territorios antes inaccesibles. Pueden diseñar anticuerpos contra dianas proteicas que han sido consideradas «inabordables» con los métodos convencionales, abriendo nuevas vías para tratar enfermedades que actualmente no tienen cura. La capacidad de especificar la arquitectura exacta permite crear formatos de anticuerpos más complejos, como los anticuerpos biespecíficos, que pueden unirse a dos dianas diferentes simultáneamente, una proeza de ingeniería molecular con un enorme potencial terapéutico.
Del silicio al laboratorio: el largo camino de la validación
A pesar del entusiasmo justificado, es fundamental inyectar una dosis de realismo. Una molécula diseñada en un ordenador, por muy perfecta que parezca en la simulación, no es un fármaco. Es, en el mejor de los casos, una hipótesis prometedora. El viaje desde el diseño digital hasta el paciente sigue siendo largo y está lleno de obstáculos.
El primer gran desafío es la «sintesibilidad». El hecho de que un modelo de IA pueda concebir una estructura proteica no garantiza que pueda ser producida en el mundo real por un químico en un laboratorio o a escala industrial. Los químicos y biólogos sintéticos están trabajando en el desarrollo de herramientas de IA paralelas que puedan evaluar la viabilidad sintética de un diseño, asignando una «puntuación de accesibilidad sintética» para filtrar los diseños que son pura fantasía computacional de aquellos que tienen una oportunidad real de ser creados.
El segundo y más importante obstáculo es la validación experimental. Una vez que se selecciona un candidato prometedor y sintetizable, debe comenzar el arduo proceso de pruebas en el mundo real. Primero, las pruebas in vitro, en las que la proteína se prueba en cultivos celulares en una placa de Petri para confirmar que se une a su diana y tiene el efecto deseado. Si supera esta fase, pasa a las pruebas in vivo, generalmente en modelos animales, para evaluar su seguridad, su eficacia y cómo se comporta en un organismo vivo complejo. Solo después de superar estas fases preclínicas, un candidato puede entrar en los ensayos clínicos en humanos, un proceso de varias fases que dura años y en el que la mayoría de los candidatos acaban fracasando.
La inteligencia artificial acelera de forma espectacular el primer paso de este maratón, el de la generación de ideas. Puede aumentar drásticamente la calidad y la cantidad de los «disparos a puerta». Pero no elimina la necesidad de recorrer el resto del campo. Lo que antes era un proceso de descubrimiento de una década podría acortarse, según algunas estimaciones, en varios años, y el coste podría reducirse significativamente, pero la biología sigue siendo obstinadamente compleja, y la validación rigurosa seguirá siendo el estándar de oro indispensable para garantizar la seguridad y la eficacia.
La doble hélice de la promesa y el peligro
Como toda tecnología con el poder de transformar la sociedad, la IA generativa para el diseño molecular conlleva una profunda responsabilidad y plantea serias cuestiones éticas. La misma herramienta que puede ser utilizada para diseñar un anticuerpo que cure una enfermedad podría, en teoría, ser utilizada con fines maliciosos para diseñar una toxina o un agente biológico dañino. Este problema del «doble uso» es una preocupación creciente para la comunidad científica y los organismos de seguridad, que están debatiendo cómo establecer salvaguardas y normas de supervisión para mitigar estos riesgos sin ahogar la innovación.
También surgen cuestiones de equidad y acceso. El desarrollo y la implementación de estas sofisticadas plataformas de IA requieren una enorme inversión en potencia computacional y talento especializado. Existe el riesgo de que esta tecnología quede concentrada en manos de unas pocas grandes corporaciones farmacéuticas o países ricos, ampliando la brecha sanitaria global. La democratización de estas herramientas, a través de iniciativas de código abierto y colaboraciones académicas, será fundamental para garantizar que sus beneficios lleguen a toda la humanidad.
Finalmente, está la cuestión de la fiabilidad y los sesgos. Los modelos de IA son tan buenos como los datos con los que se entrenan. Las bases de datos de proteínas existentes, aunque vastas, pueden tener sesgos inherentes o carecer de diversidad en ciertos tipos de estructuras. Un modelo entrenado con datos sesgados puede perpetuar esos sesgos en sus diseños, limitando su creatividad o pasando por alto soluciones novedosas. Garantizar la calidad, la diversidad y la integridad de los datos de entrenamiento es un desafío constante y crucial para la robustez del campo.
Un nuevo capítulo en el libro de la vida
Nos encontramos en un momento extraordinario de la historia de la ciencia. La confluencia de la biología computacional, la ingeniería de software y la inteligencia artificial nos ha otorgado un poder que las generaciones anteriores solo podían soñar: la capacidad no solo de leer y entender el libro de la vida, sino de empezar a escribir nuevos capítulos en él. Tecnologías como AIDO y su módulo StructureDiffusion representan un cambio fundamental en nuestra relación con el mundo molecular. Estamos pasando de ser meros descubridores de las soluciones que la evolución ha forjado a lo largo de eones a convertirnos en diseñadores activos de nuevas soluciones biológicas.
Esta tecnología no reemplazará a los científicos humanos. Al contrario, los potenciará, liberándolos de las tareas más tediosas y repetitivas y proporcionándoles un instrumento de una potencia creativa sin precedentes. La IA se convierte en una musa algorítmica, una colaboradora infatigable capaz de explorar el vasto universo de las posibilidades moleculares y presentar a la intuición y el ingenio humano ideas que de otro modo permanecerían ocultas. Es un nuevo tipo de microscopio, uno que nos permite ver no solo lo que ya existe, sino también todo lo que podría llegar a ser.
El camino por delante es largo y complejo. Los desafíos técnicos, de validación y éticos son formidables. Pero la trayectoria es clara. La habilidad de diseñar a medida las máquinas moleculares de la vida promete acelerar el advenimiento de una era de medicina más precisa, personalizada y poderosa. Estamos aprendiendo a hablar el lenguaje de las proteínas con una fluidez cada vez mayor, y las conversaciones que estamos a punto de tener cambiarán el mundo.
Referencias
- GenBio.ai. (s.f.). AIDO & StructureDiffusion: The AIDO module for molecular design. https://genbio.ai/aido-structurediffusion-the-aido-module-for-molecular-design/
- Jumper, J., et al. (2021). Highly accurate protein structure prediction with AlphaFold. Nature, 596(7873), 583–589.
- Watson, J. L., et al. (2023). De novo design of protein structure and function with RFdiffusion. Nature, 620(7976), 1089–1100.
- Dallago, C., et al. (2021). The challenges of deep generative models for de novo drug design. Briefings in Bioinformatics, 22(5).
- Fleming, M. (2024). How diffusion models work. Towards Data Science.
- McKinsey & Company. (2023). The dawn of the AI-powered biotech and pharma company.
- Saldaño, T. (2024). The revolution in drug discovery: AI and generative models. News Medical.
- The Royal Society. (s.f.). Ethics of artificial intelligence in drug discovery.



